Wildau (Germany), 20. Februar 2019 – Vor einem Jahr brachte das Biotech- und Lifesciences Startup Biomes die erste DNA-basierte Darmflora-Analyse [...]

KI-gestützte Früherkennung von Typ-2-Diabetes durch Darmmikrobiom-Analyse
Durch die Untersuchung des Darmmikrobioms eröffnen sich neue Möglichkeiten zur Identifizierung und Beschreibung verschiedener Krankheiten, insbesondere im Bereich der Diabetesforschung und -früherkennung. Unsere jüngste Studie zeigt auf, wie sich das Darmmikrobiom von Personen mit Typ-2-Diabetes von gesunden Menschen unterscheidet. Dadurch eröffnen sich neue Möglichkeiten für personalisierte Prävention und präzisere therapeutische Ansätze.
Neue Horizonte in der Diabetesforschung
Typ-2-Diabetes ist eine weitverbreitete Stoffwechselerkrankung, die das Leben von Millionen Menschen beeinflusst. Da sie schwere, gesundheitliche Komplikationen verursachen kann, stellt sie eine große Herausforderung für das Gesundheitssystem dar. Etwa einer von elf Erwachsenen leidet weltweit an Diabetes, wobei 90 % davon an Typ-2-Diabetes erkrankt sind.1 Eine neue Studie hat herausgefunden, dass etwa 70 % aller Typ-2-Diabetesfälle weltweit auf eine unausgewogene Ernährung zurückzuführen sind.2 Unsere aktuellsten Forschungsergebnisse, basierend auf Darmmikrobiomdaten, bieten ein mögliches Modell zur Vorhersage von Typ-2-Diabetes und tragen zu einem tieferen Verständnis der Diabetes-assoziierten, mikrobiellen Metabolismen und Signalwege bei.
Anwendung künstlicher Intelligenz zur Früherkennung von Typ-2-Diabetes
Kurz gefasst
Mit dieser Studie zeigen wir eine Methode, bei der wir eine Art künstliche Intelligenz, sog. neuronales Netzwerk, nutzen, um zwischen gesunden Menschen und Menschen mit Typ-2-Diabetes zu unterscheiden. Dieses Modell basiert auf den Meta-Mikrobiom-Daten unserer Kund*innen.
Unser Datensatz enthält derzeit über 40.000 Daten. Diese Datenpunkte enthalten Informationen über Bakterien und ihre Funktionen, sowie weitere Daten zu Körpermaßen, Lebensstil, Krankheiten und Medikamenteneinnahme. Dieser umfangreiche Datensatz hilft uns, ein künstliches Gehirn, auch bekannt als neuronales Netzwerk, zu trainieren, um Typ-2-Diabetes zu erkennen. Diese Proof-of-Principle-Studie soll zeigen, wie eine künstliche Intelligenz mit großen Datensätzen trainiert werden kann. Dies bietet sehr interessante Möglichkeiten für die Erkennung und Erstellung von Risikoprofilen.

Es wurden Bakterien und deren Stoffwechsel-Profile aus Stuhlproben von Diabetiker*innen (Typ 2) (n=272) und gesunden Personen (n=674) durch Next-Generation-Sequenzierung von 16S rRNA analysiert. Anschließend wurden daraus bakterielle Stoffwechselwege abgeleitet.
Das Ziel war es ein neuronales Netzwerk mit den Mikrobiomdaten zu konstruieren, welches die Identifizierung von Typ-2-Diabetes ermöglicht. Das künstliche neuronale Netz wurde trainiert und es zeigt sich, dass eine Klassifizierung von Typ-2-Diabetes und gesunder Kontrollen auf der Grundlage funktioneller, mikrobieller Profile möglich ist und das mit einer Genauigkeit von 85 %. Somit konnte mit Hilfe von Machine Learning ein Vorhersagemodell für die Wahrscheinlichkeit von Typ-2-Diabetes entwickelt werden.
Einfacher ausgedrückt: Zukünftig könnte mit Hilfe von KI das Darmmikrobiom als Biomarker für diverse Erkrankungen, wie Typ-2-Diabetes, genutzt werden.
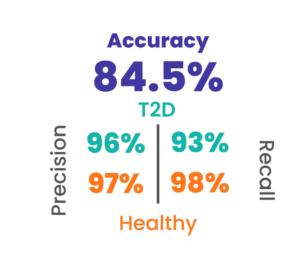
Die Studie konzentriert sich nicht nur auf die Klassifizierung mittels neuronaler Netze, sondern auch auf die Identifizierung entscheidender bakterieller Signalwege: Vor allem Stoffwechselwege der Aminosäure- , Vitamin-Biosynthese und Fermentation beeinflussen die Vorhersagbarkeit von Typ-2-Diabetes.
Diese Studie zeigt das unglaubliche Potenzial, das in unseren Darm-Mikrobiom und neuronalen Netzwerken liegt, um das Risiko von Krankheiten wie Typ-2-Diabetes vorherzusagen. Wir hoffen, dass diese Erkenntnisse in Zukunft dazu beitragen können, Risikoprofile zu erstellen und Behandlungen zu personalisieren.
Die Integration neuronaler Netzwerke in unsere Analyse des Darmmikrobioms eröffnet neue Wege für die Erkennung von Typ-2-Diabetes. Unsere Studie liefert nicht nur ein vielversprechendes Modell für die Krankheitsvorhersage, sondern verbessert auch das Verständnis der mikrobiellen Metabolismen und Signalwege, die mit Diabetes assoziiert sind. Dies bietet neue Möglichkeiten für personalisierte Therapieansätze und präziseres Krankheitsmanagement.
Isabel D., Spezialistin für Bioinformatik und Datenanalyse

Teste dein Darmmikrobiom
Bestelle jetzt die Darmfloraanalyse INTEST.pro und starte ein gesünderes Leben.


